ژنتیک پزشکی :در این پست پاسخ تست های تالیفی مرتبط با تدریس انجام شده توی کانال ژنتیک پزشکی در مورد فصل نهم ژنتیک پزشکی ایمری ارائه میشه. دوستانی که مایلیند و عضو کانال نیستن میتونن توسط این لینک عضو کانال ژنتیک پزشکی که توش ایمری برای ارشد و دکتری ژنتیک تدریس میشه، بشن و تدریسا رو هم داشته باشن.
۱- پاسخ: ج
۲- پاسخ: د- طبق تعریف بازگشت به میانگین (Regresion to the mean) و توزیع طبیعی در مورد صفات کمی، زاده های دو فرد دارای هوش پایین دارای هوش بالاتر از والدین و متمایل به میانگین هوشی جمعیت خواهد بود (هرچند که به میانگین جمعیت نمی رسد)
۳-پاسخ: ب- در مورد مدل استعداد/ آستانه (Liability/ Threshold model)) توجه به نکات زیر ضروری است:
استعداد: همه عامل هایی که توسعه ناهنجاری چند عاملی را متأثر می کنند به عنوان یک وجود واحد منفرد، استعداد یا آمادگی محسوب می شوند.
استعدادهای همه افراد، متغیر پیوسته ای را تشکیل می دهد که هم در جمعیت عمومی و هم در بستگان توزیع نرمال دارد (شکل).
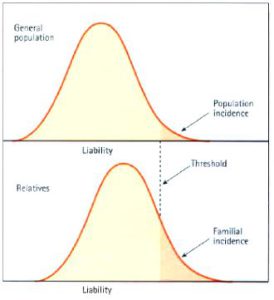
منحنی های مربوط به بستگان به سمت راست جابجا می شود که دامنه این جابجایی مستقیماً وابسته به نزدیکی رابطه آنها به مورد نمایه یا شاخص مبتلا (Index case) است (شکل ).
پیامدهای الگوی استعداد / آستانه
وضعیت در بین بستگان بیمارانی که با بیشترین شدت به بیماری مبتلا هستند بالاترین شیوع را دارد.
هرچه درجه خویشاوندی نزدیک تر باشد، شیوع بیماری بیشتر است.
چنانچه بیش از یک فرد مبتلا با خویشاوندی نزدیک وجود داشته باشد، خطرات برای بستگان دیگر افزایش می یابد.
خطر تکرار برای بستگان درجه یک، برابر جذر فراوانی جمعیت معمولی است. مثلاً اگر بروز ۱/۱۰٠٠ باشد سیب ها و فرزندان ریسکی حدود ۱/۳۲ یا ۳٠% دارند.
در صورت رایج تر بودن وضعیت در افراد با یک جنسیت ویژه، خویشاوندان فرد مبتلا با جنسیتی که کمتر مبتلا می شوند نسبت به خویشاوندان فرد مبتلا با جنسیتی که بیشتر مبتلا می شوند، در معرض خطر بیشتری قرار دارند. مثلاً در بیماری تنگی پیلور (pyloric stenosis) با نسبت ۵ به ۱ مرد به زن، نسبت فرزندان مبتلای بیماران نشانه مرد برای پسران ۵/۵% و برای دختران ۴/۲% است. در حالی که ریسک فرزندان بیماران نشانه زن برای پسران ۴/۱۹% و برای دختران ۳/۷% است.
۴-پاسخ: د
تعریف توارث پذیری: نسبتی از سبب شناسی بیماری که می توان به عامل های ژنتیکی در برابر با عامل های محیطی نسبت داد.
هر چه ارزش برای توارث پذیری بیشتر باشد، نقش عامل های ژنتیکی بیشتر خواهد بود.
با نشانه h2 توصیف می شود و با نسبتی از ۱ یا در صد بیان می شود:
h2 = (واریانس ژنتیکی)/( واریانس ژنتیکی + واریانس محیطی)= (واریانس ژنتیکی)/(واریانس فتوتیپی)
بنابراین، به عبارت دیگر توارث پذیری یعنی قابلیت پیش بینی فنوتیپ از طریق ژنوتیپ خواهد یود
۵- پاسخ: الف- جدول ۲ فصل ۹
۶- پاسخ: گزینه د-
گزینه های الف و ب و ج با توجه به توضیحات سوالات قبل کاملا صحیح هستند. در مورد گزینه د موارد زیر را به خاطر بسپارید:
وراثت پذیری و دو قلوها
وراثت پذیری را می توان از درجه شباهت یا همانندی بین خویشاوندان که در شکل ضریب همبستگی یا تطابق بیان می شود تخمین زد (Correlation Coefficient).
دوقلوهای دو تخمی امکان تطابق بیماری در محیط یکسان را فراهم می آورند.
دوقلوهای تک تخمی امکان مقایسه وابستگان با ژنوتیپ یکسان را در محیط مشترک یا غیر مشترک فراهم می آورند.
h^2= (DZ – واریانس در دوقلوهای MZ واریانس در دوقلوهای )/(DZواریانس در دوقلوهای)
بنابراین وراثت پذیری را می توان از طریق بررسی میزان تطابق دوقلوهای تک تخمکی و دوتخمکی محاسبه کرد
ارزش دوقلوها در مطالعات ژنتیک انسانی توسط فرانسیس گالتون مطرح شد.
تشابه بالاتر در صفات ژنتیکی در دوقلوهای MZ (تک تخمکی )نسبت به دوقلوهای DZ (دو تخمکی) همواره یک تأثیر ژنتیکی را اثبات نمی کند، چرا که همه دوقلوهای MZ از یک جنس و نیمی از دوقلوهای DZ از جنس های متفاوت اند.
یک آزمایش ایده آل برای جداسازی اثرات ژن ها و محیط مشترک مطالعه دوقلوهای MZی است که در زمان تولد از هم جدا شده-اند که البته این تحقیقات دارای نقطه ضعف هایی است:
وجود تعداد کمی از این افراد
جدایی ناکامل
علاقه به مطالعه روی دوقلوهای جداشده فوق العاده شبیه
عدم تشخیص علل محیطی درون رحمی از علل ژنتیکی. مثلاً طبق تحقیقات هورمون های مادری ممکن است بر جنین درون رحم و در نتیجه جهت گیری جنسی (ژن مربوط به هم جنس گرایی) آینده او اثرگذار باشد.
۷- پاسخ: ب-
به دلیل طبیعت پیچیده بیماری های چند عاملی یا پیچیده و وجود واریانت های ژنتیکی با اثر کم که با همدیگر و محیط برهمکنش می کنند اغلب مطالعات پیوستگی برای آشکارسازی این اثرات شکست می خورند و روش مطالعه همراهی روش قدرتمندتری برای یافتن واریانت های ژنتیکی دخیل در بیماری های کمپلکس است. برای بررسی روش های مختلف اطلاعات زیر مناسب خواهند بود:
مطالعات شناسایی ژن های مسبب اختلالات چند عاملی
آنالیز پیوستگی (Linkage Analysis):
تجزیه و تحلیل جفت برادر– خواهر مبتلا
نقشه کشی پیوستگی ناهمسنگی (بی تعادلی، Linkage disequilibrium:LD)
همراهی یا همباشی یا وابستگی (Association):
آزمون ناهمسنگی انتقال (TDT)
همراهی ژنوم کامل:
آزمون همزمان SNP (Single Nucleotide Polymorphism)
آنالیز پیوستگی
ابتدا به مفهوم آنالیز تفکیک (Segregation anaysis) می پردازیم:
آنالیز تفکیک، یک ابزار آماری برای آنالیز وراثت هر نوع صفت است.
در بیماری های چندعاملی، هم فاکتورهای ژنتیکی (شامل پلی ژنی، الیگوژنی یا مونوژنی، مندلی، با هر نوع وراثتی) و هم فاکتورهای محیطی (شامل متغیرهای خانوادگی و غیرخانوادگی) درگیر هستند. در آنالیز پیچیده تفکیک، مجموعه کاملی از الگوهای وراثتی ممکن، فراوانی های ژنی، نفوذ و… توسط آنالیز کامپیوتری مدلسازی می شوند. سپس عملکرد هر مدل با این مدل مخلوط مقایسه می شود و نزدیک ترین مدل در تفسیر اطلاعات به مدل مخلوط مدنظر قرار می گیرد.
ناهمگنی ژنتیکی شدید اکثر بیماری های چندعاملی موجب ضعف آنالیز تفکیک شده است. در نتیجه مطالعات به سمت آنالیز پیوستگی و همراهی (ارتباط) معطوف شده است.
آنالیز پیوستگی صفات چندعاملی
در آنالیز پیوستگی صفات چند عاملی، آنالیز استاندارد lod به دلیل نیاز به یک مدل ژنتیکی دقیق نمی تواند مورد استفاده قرار گیرد. به منظور حل این مشکل از خانواده های نسبتاً مندلی “near-Mandelian” استفاده می شود. در این مورد پارامترهای یک مدل ژنتیکی (نوع وراثت، فراوانی ژنی و میزان نفوذ هر ژنوتیپ) بوسیله آنالیز تفکیک تعیین می شود و سپس در یک آنالیز پیوستگی استاندارد (پارامتریک) به کار می رود. از نقاط ضعف این روش می توان به موارد زیر اشاره نمود:
*ناهمگنی بیماری چندعاملی
*بروز بیماری هایی که در آنها به طور اتفاقی بسیاری از فاکتورهای تعیین کننده بیماری همواره در اکثر افراد در این خانواده نسبتاً مندلی وجود دارد.
*الگوی نسبتاً مندلی کاذب
در نتیجه از روش های غیرپارامتریک یا فاقد مدل یا فارغ از الگو (free model) آنالیز پیوستگی استفاده می شود که به دنبال آلل ها یا قطعات کروموزومی مشترک با اشتراک بالاتر از نسبت های تصادفی حاصل از تفکیک مندلی در افراد مبتلا هستند. در این روش قطعاتی که از نظر تبار همسان هستند (IBD) یعنی از یک جد مشترک به ارث رسیده اند (در مقابل قطعات همسان از نظر وضعیت (IBS)) در نظر گرفته می شود. در تعیین IBD میکروستلایت های چند آللی کارامدتر از مارکرهای دو آللی (مانندSNP) هستند.
شکلی از آنالیز پیوستگی غیر پارامتریک آنالیز جفت خواهر- برادر (سیب) مبتلا (ASPs) است. آنالیز چند لوکوسی به آنالیز تک لوکوسی ارجحیت دارد و برنامه کامپیوتری MapMarker/sibs برای آنالیز اطلاعات چند نقطه ای ASP به کار می رود.
برنامه هایی مانند Gene hunter برای آنالیز قطعه مشترک در مورد سایر نسبت های خویشاوندی به کار می رود.
نقاط ضعف آنالیز APS:
*شناسایی نواحی کاندید بزرگ که احتمالاً دارای ژن های بسیاری هستند.
*عدم بررسی مارکرها به لحاظ میزان نزدیکی و تعیین حد نوترکیبی (برخلاف نقشه یابی مندلی)
نقشه کشی عدم تعادل پیوستگی (LD= Linkage Disequilibrium)
پس از تشخیص یک قطعه کروموزومی دخیل در استعداد به یک بیماری چندعاملی، بوسیله نقشه کشی ظریف و دقیق (Fine mapping) قطعه مذکور را کوچکتر می کنند. در قوی ترین روش، از نقشه کشی عدم تعادل پیوستگی استفاده می شود که به موجب آن ژنوتیپ SNP های واقع در درون قطعه برای تهیه هاپلوتایپ ها تعیین می شود. نقاط رخداد کراسینگ اور با تعیین بلوک های LD، قطعه ژنتیکی را کوتاهتر می کند.
سپس با تعیین توالی ژن های کاندید در ناحیه، واریانت های DNA از نظر همراهی با بیماری بررسی می شوند. بنابراین LD را می توان همراهی آللی نامید (هاپلوتایپ، مجموعه ای از آلل های موجود در لوکوس های متصل بر روی یک کروموزوم واحد است). LD یک مقدار آماری درباره جمعیت ها است (نه افراد).
هاپلوتایپ های درون یک جمعیت باید توسط کمترین تعداد نوترکیب ها با یکدیگر خویشاوند باشند. LD تنها به فاکتورهای حساسیتی اشاره دارد که از اجداد مشترک قدیمی به ارث رسیده اند. با در نظر گرفتن کسر نوترکیبی θ برای یک مارکر با لوکوس بیماری مشترک، بعد از n میوز (n نسل)، تعداد 〖۱-θ〗^n از کروموزوم ها همراهی این مارکر با لوکوس را حفظ خواهند کرد.
دلایل انتخاب SNP ها به عنوان مارکر
اندازه فراوان آن ها که امکان بررسی قطعات کروموزومی بسیار کوتاه را فراهم کرده است.
میزان رخداد کمتر جهش در مقایسه با STR ها که بخصوص زمانی اهمیت می یابد که هدف شناسایی قطعات کروموزومی حفظ شده قدیمی باشد.
تعیین آسان ژنوتیپ SNP ها در مقیاس وسیع
انحرافات یا بی قاعدگی هایی در مطالعات LD مشاهده شد که دلایل آن می تواند اثرات ترکیب شده چند فاکتور باشد:
الف) نوترکیبی: وجود نقطه داغ بین SNP های مجاور موجب مشاهده کاهش یا فقدان LD می شود و فقدان نقطه داغ در یک لوکوس بزرگ موجب شدت LD می شود.
ب) تبدیل ژنی (Gene conversion): در صورت جایگزین شدن یک بخش درونی کوچک از قطعه، باز هم مارکرهای موجود در دو سمت قطعه با یکدیگر LD نشان می دهند.
ج) تاریخچه جمعیت: هرچه جمعیت قدیمی تر باشد، قطعات حفظ شده کوتاهتر است. LD در جمعیت هایی که در اندازه ثابت باقی مانده-اند نسبت به جمعیت هایی که به تازگی دستخوش توسعه شده اند دامنه کوتاهتری دارد.
با توجه به تعداد زیاد SNP ها در هر قطعه کروموزومی تعداد هاپلوتایپ های احتمالی بسیار زیاد خواهند بود. در واقعیت، تعداد کمی از این هاپلوتایپ ها اکثر کپی های یک کروموزوم را در جمعیت توجیه می کند. برای شناسایی چند هاپلوتایپ می توان تنها تعداد کمی از SNP ها (tag- SNPs) را تعیین ژنوتیپ نمود.
۲٫ مطالعه ارتباط یا همراهی یا همباشی (Association)
همراهی رخداد یک آلل ویژه در گروهی از بیماران است که بیشتر از آنچه که توسط شانس صورت می گیرد رخ می دهد.
همراهی به جستجوی فراوانی تجمع آلل های خاص در افراد بیمار در مقایسه با افراد سالم می پردازد.
شناسایی یک جایگاه مستعد به مفهوم آن نیست که ژن بیماری به طور قطع تعیین هویت گردیده است مثلاً تنها ۱ درصد افراد HLA B27 به بیماری اسپوند لیتیس انکلوزینگ مبتلا هستند.
(Relative Risk)RR = خطر نسبی
مطالعات همراهی آسان تر از آنالیز پیوستگی است و در شناسایی آلل های با حساسیت ضعیف قدرتمندتر از پیوستگی عمل می کند.
احتمال خطر نسبی ۱ به این معنی است که مثبت بودن برای داشتن یک آلل خاص هیچ احتمال خطر بیشتری را به بیماری نمی دهد.
مزیت نسبت احتمال این است که مستقیماً از روی نتایج مطالعه بیمار- کنترل (Case- Control) بدون نیاز به اطلاع در مورد میزان بروز در جمعیت محاسبه می شود.
اکثر مطالعات همراهی با بیماری، کشف ارتباطی است که بواسطه LD ایجاد می شود.
علل یک همراهی در جمعیت
علت مستقیم: داشتن آللA احتمال ابتلا به بیماری D را افزایش می دهد اما برای توسعه بیماری در فرد نه لازم و نه کافی است.
اثر اپی ستاتیک: افراد بیمار در صورت داشتن آلل A به احتمال بیشتری زنده مانده و صاحب فرزند می شوند.
قشربندی (لایه لایه شدن) جمعیت: جمعیت دارای چندین زیر جمعیت مجزا از نظر ژنتیکی (بدون interbreedingآزادانه) است و در چنین جمعیتی به طور اتفاقی بیماری D و آلل A هر دو در یک زیر جمعیت فراوان اند. مطالعات همراهی نسبت به طبقه بندی های مختلف جمعیت حساسیت دارد.
LD : آلل A وابسته به بیماری نشان دهنده قطعه ای از کروموزوم جَدی است که با داشتن یک نوع توالی سبب حساسیت به بیماری می شود.
آزمون ناهمسنگی انتقال (TDT)
راهی برای غلبه بر مشکلات طبقه بندی جمعیت است.
نیازمند گروه های سه نفری شامل یک فرزند مبتلا و هر دو والد هتروزیگوس است.
تعداد دفعات انتقال آلل مورد پرسش به فرزندان مبتلا و تعداد دفعات انتقال دیگر آلل ها را مقایسه می کند: انتقال بیشتر آلل مورد پرسش موجب احتمال بیشتر همراهی می شود.
هم اکنون مطالعات مرسوم بیمار– کنترل (به شرط سازگاری بیماران و کنترل ها) به عنوان جایگزین TDT انجام می گیرد. زیرا نسبت به TDT 50% به نمونه های کمتری نیاز دارد و در بیماری های دیر شروع شونده که والدین به ندرت در دسترس هستند عملی تر است.
ناهمگنی آللی و ناهمگنی لوکوسی به ترتیب کارایی یک آزمون همراهی و پیوستگی را از بین می برد.
پیوستگی ارتباط بین لوکوس هاست(ارتباط ژنتیکی) ولی همراهی رابطه بین آلل ها و یا فنوتیپ های خاص است (یک مشاهده آماری). حال اگر خانواده و جمعیت در هم ادغام شوند پیوستگی و همراهی هر دو عمل می کنند.
همراهی کل ژنوم:
در روش ژن کاندید تنها همراهی هایی بدست می آیند که تا حد زیادی تکرار می شوند. راهکارهایی امکان بررسی مطالعات همراهی را در سطح ژنوم فراهم کرده اند:
پروژه Hap Map: با وجود ۱۰ میلیون SNP در ژنوم انسان و مشاهده LD بین تعداد زیادی از SNP ها، این پروژه فراوانی SNP ها و هاپلوتایپ ها را در جمعیت های مختلف تعیین می کند. با تعیین ژنوتیپ حدود۵۰۰ هزار SNP در اکثر جمعیت ها اطلاعات مربوط به کل SNP های معمول ژنوم انسان بدست می آید (با حداقل فراوانی آللی> 5%) در جمعیت های آفریقایی به دلیل پایین بودن LD کلی، تعدادSNP های لازم برای تعیین ژنوتیپ یک میلیون است.
۲- مطالعات همراهی ژنوم کامل(GWA=Genome Wide Association): در این مطالعات به جای یک واریانت ، مقایسه واریانت ها در کل ژنوم به شکل مطالعات Case – control انجام می شود. در یک مطالعه GWA معمول، ۵۰۰ هزار تا یک میلیون SNP با استفاده از ریزآرایه SNP (SNP chip) تعیین ژنوتیپ می شوند. برخلاف روش ژن کاندید، مطالعه GWA نیازی به فرضیه ندارد. در مطالعه آماری همراهی، ارزش ۰۵/۰ p< به عنوان عدم احتمال وقوع تصادفی در نظر گرفته می شود و این ارزش در مطالعهGWA 8- P=5×۱۰(آستانه پذیرفته شده پایین تر از همراهی مثبت کاذب) است، چرا که با افزایش تعدادSNP های مورد آزمایش آستانه ارزش P نیز باید تغییر کند (۱/۲٠ آزمایشات به طور تصادفی ارزشی کمتر از ۰۵/۰ p< نشان می دهند).
۳- پروژه هزار ژنوم (Thousand Genomes Project): در این پروژه نقشه دقیق آلل ها با فراوانی ۱% بدست خواهد آمد و علاوه بر SNP ها، سایر انواع واریانت ها نظیر تنوع تعداد نسخه (CNV) فراهم خواهد شد.
۸- پاسخ: د-
این نکات را در مورد وراثت پلی ژنیک و توزیع نرمال بخاطر بسپارید:
آشنایی با مفاهیم چند اصطلاح
صفات کیفی یا منفصل یا مونوژنیک (Qualitative traits):
قابل دسته بندی بوده و فرد می تواند صفت را بروز دهد یا ندهد.
صفات کمی یا پیوسته یا پلی ژنیک(Quantitative traits):
چندین ژن نقش دارند و هر یک، تأثیر افزایشی جزئی اعمال می کنند- رابطه غالب و مغلوبی ندارند- قابل اندازه گیری اند.
توارث چند عاملی یا پیچیده (Multifactorial or Complex Inheritance):
نتیجه تعامل پیچیده وراثت پلی ژنیک و عوامل محیطی است.
وراثت پلی ژنیک و توزیع نرمال
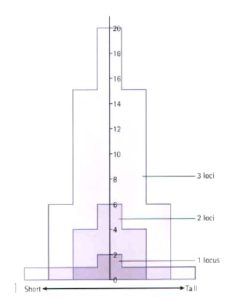
تفاوت افراد در صفات پیوسته جزیی است.
نمودار تعداد افراد (y) در مقابل مقدار صفت (x)، منحنی زنگوله شکل (Gaussian) توزیع نرمال را پدید می آورد.
هر چه تعداد ژن های دخیل در ایجاد صفت بیشتر باشد منحنی به حالت توزیع نرمال نزدیک تر می شود.
مثالهایی از صفات پلی ژنیک با توزیع نرمال
فشار خون،کف شناسی (شمارش برآمدگی خطی)، دور سر، قد، هوش،رنگ پوست
نمودار صفات پیوسته و نقش تعداد ژنها
هر لوکوس دارای ۲ آلل با فراوانی یکسان است.
در پایان به دانشجویانی که تمایل دارند از مباحثی که از ژنتیک پزشکی ایمری در کانال ژنتیک پزشکی متعلق به همین سایت تدریس می شود استفاده کنند پیشنهاد می گردد از طریق این لینک به کانال ژنتیک پزشکی بپیوندند.